Rの関数から brm {brms} を確認します。
本ポストは、こちらの続きです。

brm関数について
brmは、Rのbrmsパッケージで用意されている関数です。
「Bayesian Regression Models using Stan」の名の通り、統計モデリング言語Stanを用いてベイズモデルを構築・推定します。
本関数の特徴は、rstanarmパッケージがglmやlmerといった既存のR関数と似た使いやすさを提供するのに対し、brmsはより複雑なモデル構造にも対応できる、LME4風のformula構文を採用している点です。
brm関数を用いることで、以下のような様々なモデルを、統一的なインターフェースで扱うことができます。
- 一般化線形モデル (GLM)
- 一般化線形混合効果モデル (GLMM) / 階層モデル
- 非線形モデルおよび階層非線形モデル
- 生存時間分析モデル
- 時系列モデル
- 測定誤差モデル
- ゼロ過剰(zero-inflated)モデルやハードルモデル
- 多変量モデル(複数の目的変数を同時に扱う)
brmsは、ユーザーが指定したformula構文を解釈し、背後で自動的にStanのコードを生成・コンパイルしてMCMCサンプリングを実行します。
それゆえ、ユーザーはStanのコードを直接書くことなく、Stanが持つサンプリング能力を利用することができます。
brm関数の主要な引数
以下に、brm関数の主要な引数について確認します。
library(brms)
args(brm)function (formula, data, family = gaussian(), prior = NULL, autocor = NULL,
data2 = NULL, cov_ranef = NULL, sample_prior = "no", sparse = NULL,
knots = NULL, drop_unused_levels = TRUE, stanvars = NULL,
stan_funs = NULL, fit = NA, save_pars = getOption("brms.save_pars",
NULL), save_ranef = NULL, save_mevars = NULL, save_all_pars = NULL,
init = NULL, inits = NULL, chains = 4, iter = getOption("brms.iter",
2000), warmup = floor(iter/2), thin = 1, cores = getOption("mc.cores",
1), threads = getOption("brms.threads", NULL), opencl = getOption("brms.opencl",
NULL), normalize = getOption("brms.normalize", TRUE),
control = NULL, algorithm = getOption("brms.algorithm", "sampling"),
backend = getOption("brms.backend", "rstan"), future = getOption("future",
FALSE), silent = 1, seed = NA, save_model = NULL, stan_model_args = list(),
file = NULL, file_compress = TRUE, file_refit = getOption("brms.file_refit",
"never"), empty = FALSE, rename = TRUE, ...)
NULLformula:モデルの構造を定義する
brmsformulaオブジェクトです。線形モデル:
y ~ x1 + (1 | group)のように、lme4パッケージと類似した構文でランダム効果(変量効果)を指定できます。非線形モデル:
bf(y ~ eta, eta ~ x, nl = TRUE)のように、bf()関数を用いて非線形な関係を定義します。パラメータ自体を他の変数で予測する、といった複雑な構造も記述可能です。分布モデル:
bf(y ~ x, sigma ~ x)のように、目的変数の標準偏差sigmaなどの分布パラメータも変数でモデル化できます。
data:モデルで使用するデータが含まれたデータフレームを指定します。
family:目的変数の確率分布とリンク関数を指定します。
gaussian,poisson,binomial,Gammaなどに加え、student,beta,zero_inflated_poissonなど、glmよりも多くの分布ファミリーが利用可能です。prior:モデル内の全てのパラメータ(回帰係数、ランダム効果の標準偏差、切片、誤差項など)に対して、事前分布を明示的に指定します。
set_prior()またはprior()関数を用いて、prior(normal(0, 1), class = "b")のように、パラメータのクラス (class) を指定して設定します。chains,iter,warmup,cores:MCMCサンプリングを制御するための引数です。
-
chains: 実行するマルコフ連鎖の数。 -
iter: 1チェーンあたりの総サンプリング回数。 -
warmup: パラメータの事後分布に収束するための準備期間(バーンイン)。ここで得られたサンプルは最終的な結果から破棄されます。 -
cores: 計算に使用するCPUコア数。chains数と合わせることで、並列計算により実行時間を短縮できます。
-
control:Stanのサンプラーの挙動をより詳細に制御するためのリストを指定します。例えば、サンプリングの発散(divergence)が多い場合には
control = list(adapt_delta = 0.99)のように設定し、サンプラーのステップサイズを調整することがあります。backend:Stanを実行するためのバックエンドを指定します。デフォルトは
"rstan"ですが、"cmdstanr"を指定することも可能です。
シミュレーション
ここでは、「個体(group)ごとに成長パターンが異なる、植物の成長曲線」をシミュレーションの題材とします。
このデータが「階層非線形モデル」となる理由:
非線形性:
植物の成長は、初期は緩やかで、ある時期に急成長し、やがて飽和(頭打ち)に達するという、典型的なS字(シグモイド)カーブを描きます。このような関係は、直線では表現できない非線形な関係です。
階層性:
栽培環境や遺伝的な違いにより、植物の個体ごとに「最終的な大きさ」や「成長の速さ」は異なります。このように、データが個体などのグループ構造を持ち、パラメータがグループごとに変動する場合、それは階層(マルチレベル)構造となります。
この2つの特徴を組み合わせたものが、階層非線形モデルです。
以下に、シミュレーションを実行するためのRコードを示します。ロジスティック成長曲線 y = Asym / (1 + exp(-(x - xmid) / scal)) を非線形関数として使用します。
stan_output <- "D:/stan_output"
# 必要なパッケージを読み込みます
library(ggplot2)
library(dplyr)
library(tidyr)
# シミュレーションのための乱数シードを設定します
seed <- 20251102
set.seed(seed)
# 1. サンプルデータの作成
# 個体数(グループ数)
n_groups <- 10
# 各個体の観測点数
n_obs_per_group <- 15
# --- 真のパラメータ(集団レベル)を設定します ---
# 成長曲線のパラメータの平均値を定義します
# Asym: 最終的な成長の飽和点(漸近線)
pop_asym <- 100
# xmid: 成長率が最大になる時点(変曲点)
pop_xmid <- 8
# scal: 成長の速さ(値が小さいほど急成長)
pop_scal <- 1.5
# 観測誤差の標準偏差
pop_sigma <- 5
# --- 真のパラメータ(個体レベルのばらつき)を設定します ---
# 各パラメータの個体差(ランダム効果)の標準偏差を定義します
sd_asym <- 20
sd_xmid <- 2
# 各個体のパラメータを生成します
group_params <- tibble(
group = 1:n_groups,
# 平均pop_asym、標準偏差sd_asymの正規分布から個体ごとのAsymを生成
asym = rnorm(n_groups, mean = pop_asym, sd = sd_asym),
# 平均pop_xmid、標準偏差sd_xmidの正規分布から個体ごとのxmidを生成
xmid = rnorm(n_groups, mean = pop_xmid, sd = sd_xmid)
)
# 上記のパラメータに基づいて観測データを生成します
sim_data <- tibble(
# 1~10のグループIDを各15回繰り返す
group = rep(1:n_groups, each = n_obs_per_group),
# 観測時点(例:日数)
day = rep(1:n_obs_per_group, times = n_groups)
) %>%
# 各個体のパラメータをデータに結合
left_join(group_params, by = "group") %>%
# 成長曲線に従うyの期待値を計算
mutate(
mu = asym / (1 + exp(-(day - xmid) / pop_scal)),
# 期待値muに観測誤差(正規分布ノイズ)を加えて最終的な身長を生成
height = rnorm(n(), mean = mu, sd = pop_sigma)
)
cat("--- 作成されたサンプルデータの一部を確認 ---\n")
print(str(sim_data, 10))--- 作成されたサンプルデータの一部を確認 ---
tibble [150 × 6] (S3: tbl_df/tbl/data.frame)
$ group : int [1:150] 1 1 1 1 1 1 1 1 1 1 ...
$ day : int [1:150] 1 2 3 4 5 6 7 8 9 10 ...
$ asym : num [1:150] 98.4 98.4 98.4 98.4 98.4 ...
$ xmid : num [1:150] 5.37 5.37 5.37 5.37 5.37 ...
$ mu : num [1:150] 5.05 9.39 16.77 28.12 43.09 ...
$ height: num [1:150] 3.28 16.23 28.03 21.51 43.36 ...
NULL# 2. brmによる階層非線形モデルの推定
# --- 事前分布を設定します ---
priors <- c(
# Asymとxmidの集団レベル平均に対する事前分布
prior(normal(100, 30), nlpar = "Asym", class = "b"),
prior(normal(8, 5), nlpar = "xmid", class = "b"),
# scalは正の値しか取らないため、対数正規分布を設定し、下限(lower bound)が0であることを明記します
prior(lognormal(log(1.5), 1), nlpar = "scal", class = "b", lb = 0),
# 観測誤差の標準偏差に対する事前分布(t分布)
prior(student_t(3, 0, 10), class = "sigma"),
# ランダム効果の標準偏差に対する事前分布(t分布)
prior(student_t(3, 0, 10), class = "sd", nlpar = "Asym"),
prior(student_t(3, 0, 10), class = "sd", nlpar = "xmid")
)
cat("--- 事前分布の確認 ---\n")
print(priors)
# --- モデル式を定義します ---
# bf()を用いて非線形モデル式と、各パラメータのモデル式を定義します
# Asymとxmidが個体(group)ごとに変動する(ランダム効果を持つ)ことを指定します
nonlinear_formula <- bf(
height ~ Asym / (1 + exp(-(day - xmid) / scal)),
Asym ~ 1 + (1 | group),
xmid ~ 1 + (1 | group),
scal ~ 1,
nl = TRUE
)
cat("\n--- モデル式の確認 ---\n")
print(nonlinear_formula)--- 事前分布の確認 ---
prior class coef group resp dpar nlpar lb ub tag source
normal(100, 30) b Asym <NA> <NA> user
normal(8, 5) b xmid <NA> <NA> user
lognormal(log(1.5), 1) b scal 0 <NA> user
student_t(3, 0, 10) sigma <NA> <NA> user
student_t(3, 0, 10) sd Asym <NA> <NA> user
student_t(3, 0, 10) sd xmid <NA> <NA> user
--- モデル式の確認 ---
height ~ Asym/(1 + exp(-(day - xmid)/scal))
Asym ~ 1 + (1 | group)
xmid ~ 1 + (1 | group)
scal ~ 1# --- brm関数でモデルを推定します ---
# MCMCの進捗表示を抑制するために refresh = 0 を指定します
fit_brm_nl <- brm(
formula = nonlinear_formula,
data = sim_data,
family = gaussian(),
prior = priors,
chains = 4,
iter = 2000,
warmup = 1000,
cores = 4,
seed = seed,
refresh = 0
)
# stanfit オブジェクトの保存
setwd(stan_output)
saveRDS(object = fit_brm_nl, file = "fit_brm_nl.rds")# stanfit オブジェクトの読み込み
setwd(stan_output)
fit_brm_nl <- readRDS("fit_brm_nl.rds")
# 3. 結果の要約と解釈
cat("--- モデル推定結果の要約 ---\n")
print(fit_brm_nl)--- モデル推定結果の要約 ---
Family: gaussian
Links: mu = identity
Formula: height ~ Asym/(1 + exp(-(day - xmid)/scal))
Asym ~ 1 + (1 | group)
xmid ~ 1 + (1 | group)
scal ~ 1
Data: sim_data (Number of observations: 150)
Draws: 4 chains, each with iter = 2000; warmup = 1000; thin = 1;
total post-warmup draws = 4000
Multilevel Hyperparameters:
~group (Number of levels: 10)
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sd(Asym_Intercept) 12.11 3.30 7.21 20.25 1.00 1348 1941
sd(xmid_Intercept) 3.00 0.87 1.84 5.09 1.01 995 1685
Regression Coefficients:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
Asym_Intercept 98.98 4.24 90.76 107.46 1.00 1277 1817
xmid_Intercept 8.45 0.96 6.53 10.41 1.00 1072 1368
scal_Intercept 1.46 0.06 1.34 1.58 1.00 3846 2803
Further Distributional Parameters:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
sigma 5.36 0.33 4.77 6.04 1.00 3804 2812
Draws were sampled using sampling(NUTS). For each parameter, Bulk_ESS
and Tail_ESS are effective sample size measures, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).出力は、構築された階層非線形モデルの推定結果を包括的に要約したものです。主に4つのセクションから構成されています。
モデルの基本情報:
どのようなモデル式、データ、分布ファミリーが使用されたかの概要。
Multilevel Hyperparameters:
グループ(個体)間のばらつき(ランダム効果)に関する推定結果。
Regression Coefficients:
集団全体の平均的な傾向(固定効果)に関する推定結果。
Further Distributional Parameters:
観測誤差など、その他のパラメータに関する推定結果。
加えて、各推定値にはMCMCサンプリングの診断指標が付随しています。
1. モデルの基本情報
このセクションは、実行された分析の設計図を示します。
Family: gaussian:目的変数
heightの分布として正規分布(ガウス分布)を仮定していることを示します。Links: mu = identity:リンク関数が
identity(恒等)であることを意味し、モデルが身長の期待値muを直接予測することを示します。Formula:このモデルの構造を定義する部分です。
height ~ Asym/(1 + exp(-(day - xmid)/scal)):モデルの非線形な部分で、身長
heightがロジスティック成長曲線に従うことを定義しています。Asym、xmid、scalがこの曲線の形状を決めるパラメータです。Asym ~ 1 + (1 | group):パラメータ
Asym(最終的な身長)が、集団全体の平均(1で示される切片)と、個体groupごとのばらつき((1 | group)で示されるランダム効果)によって決まることを示しています。xmid ~ 1 + (1 | group):同様に、パラメータ
xmid(成長の変曲点)も、集団平均と個体ごとのばらつきを持つことを示します。scal ~ 1:パラメータ
scal(成長の速さ)は、個体差を考慮しない単一の集団平均値としてモデル化されていることを示します。
Data:使用されたデータフレーム sim_data と、観測数150であることがわかります。Draws:MCMCサンプリングの設定です。4つのチェーンで、ウォームアップ後に各チェーン1000サンプル、合計で4000の事後分布サンプルが得られたことを示します。
2. Multilevel Hyperparameters (グループ(個体)レベルの効果)
このセクションは、モデルの階層構造、つまり、個体差の大きさを定量化したものです。「ハイパーパラメータ」とは、パラメータの分布を制御するパラメータを意味します。
sd(Asym_Intercept):個体ごとの
Asym(最終的な身長)が、集団平均の周りでどの程度ばらついているかを示す標準偏差の推定値です。Estimateが12.11であり、シミュレーションで設定した真の値(sd_asym = 20)をその信用区間(7.21 – 20.25)内に捉えています。sd(xmid_Intercept):個体ごとの
xmid(成長のタイミング)のばらつきの標準偏差です。Estimateが3.00であり、こちらも真の値(sd_xmid = 2)を信用区間(1.84 – 5.09)内に捉えています。
3. Regression Coefficients (集団レベルの効果)
一般的に固定効果(Fixed Effects)と呼ばれる部分で、集団全体の平均的な成長パターンを示します。
Asym_Intercept:集団全体で平均した
Asym(最終的な身長)の推定値です。Estimateが98.98であり、真の値(pop_asym = 100)に近い値が推定されています。xmid_Intercept:集団全体で平均した
xmid(成長のタイミング)の推定値です。Estimateが8.45であり、真の値(pop_xmid = 8)を信用区間内に捉えています。scal_Intercept:集団全体で共通の
scal(成長の速さ)の推定値です。Estimateが1.46であり、真の値(pop_scal = 1.5)とほぼ一致しています。
4. Further Distributional Parameters (その他の分布パラメータ)
上記の構造では説明しきれない、データのばらつき(ノイズ)に関するパラメータです。
sigma:観測誤差の標準偏差の推定値です。個々のデータ点が、モデルが予測する成長曲線から平均してどの程度離れているかを示します。
Estimateが5.36であり、真の値(pop_sigma = 5)をほぼ再現しています。
MCMC診断指標
各パラメータの右側にあるRhat, Bulk_ESS, Tail_ESSは、推定結果の信頼性を担保するための診断指標です。
Rhat:すべてのパラメータで1.00または1.01となっており、MCMCのチェーンが正しく同じ分布に収束したことを示しています。
Bulk_ESS,Tail_ESS:実効サンプルサイズを示します。値が大きいほど、より安定した推定ができていることを意味し、今回の結果に問題は見られません。
# 4. モデルの予測結果の可視化
# --- 集団レベルの平均的な成長曲線を描画します ---
me_plot <- conditional_effects(fit_brm_nl)
plot_pop_fit <- plot(me_plot, plot = FALSE)[[1]] +
geom_point(
data = sim_data,
aes(x = day, y = height),
alpha = 0.3,
inherit.aes = FALSE
) +
labs(
title = "集団レベルでの平均的な成長曲線と95%信用区間",
x = "日数",
y = "身長"
) + theme_bw()
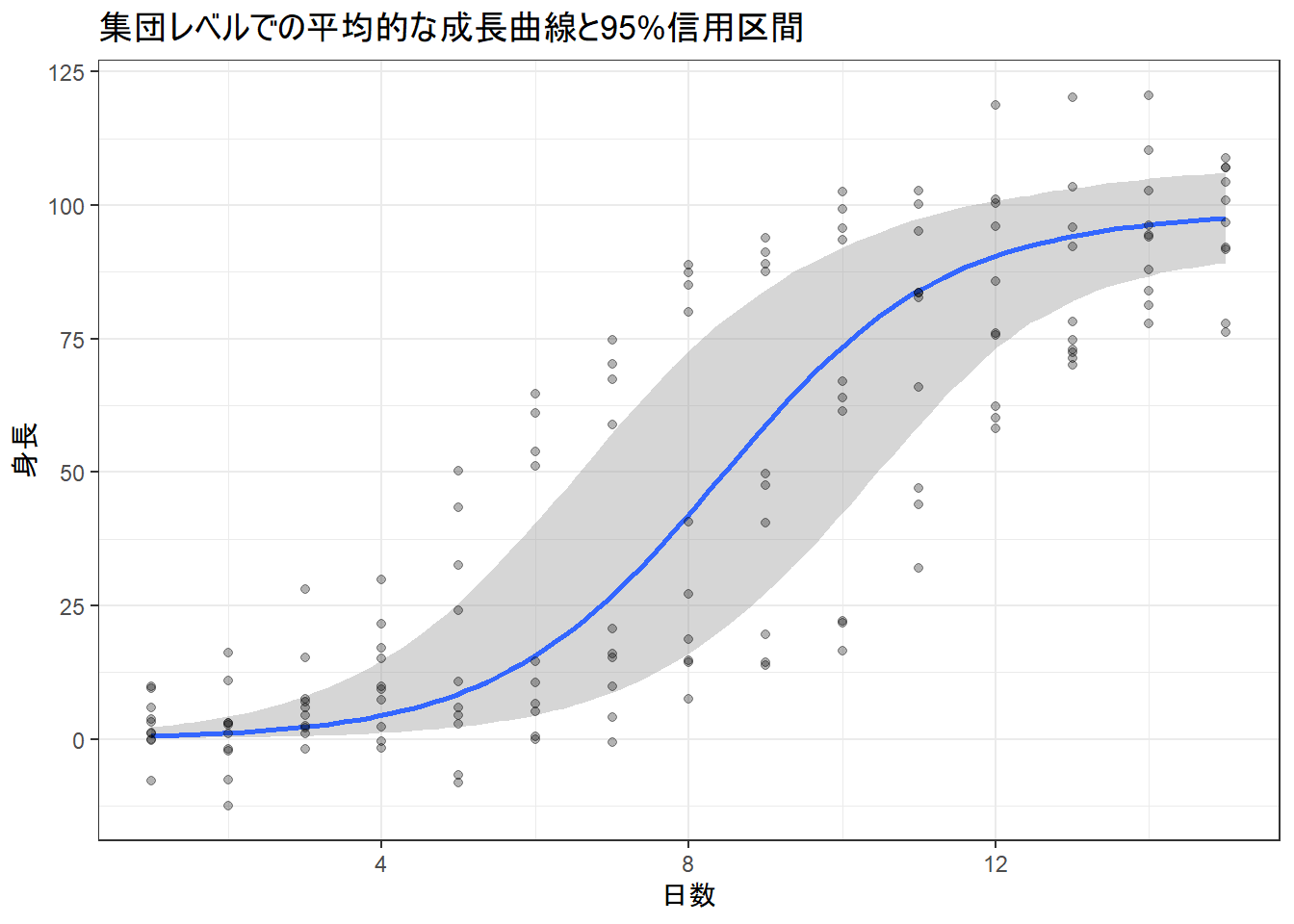
print(plot_pop_fit)Figure 1 は、構築した階層非線形モデルが、個体差をならした(平均化した)上で、この植物集団全体の平均的な成長パターンをどのように捉えているかを視覚化するものです。
個々の植物がどのように成長するかではなく、「この品種の植物は、平均的に見れば、このようなS字カーブを描いて成長する」という、集団レベルでの大きなトレンドを明らかにすることを目的とします。
1. 黒色の点 (観測データ)
- シミュレーションで作成したすべての個体(10個体分)の、すべての観測データを一つのグラフ上にプロットしたものです。
- 個体差(最終的な身長や成長タイミングの違い)が大きいため、データは全体的に広くばらついて見えます。
2. 青色の実線 (平均的な成長曲線)
- モデルが推定した集団レベルでの平均的な成長曲線です。
- モデルの要約結果における
Population-Level Effects(Asym_Intercept,xmid_Intercept,scal_Intercept)の推定値に基づいて描画されています。それゆえ、特定の個体ではなく、この集団における「架空の平均的な個体」の成長予測を示しています。 - データの中心を貫く滑らかなS字状(ロジスティック曲線)を描いていることから、モデルがデータの持つ非線形なトレンドを学習していることがわかります。
3. 灰色の帯 (平均予測に対する95%信用区間)
- 平均的な成長曲線(青い線)そのものに対する不確実性を示す95%信用区間(Credible Interval)です。
-
rstanarmの例で見た「事後予測区間」とは異なります。事後予測区間が「新しいデータ点1つがどこに現れるか」の範囲を示すのに対し、この信用区間は「集団の真の平均成長曲線が、95%の確率でこの帯の範囲内に存在する」という、平均値の不確実性を示しています。それゆえ、データ全体のばらつきよりも狭い範囲となります。 - この帯の幅は、モデルが
Asym_Interceptなどの集団レベルパラメータをどの程度の確信度で推定できたかを反映しています。幅が狭いほど、平均的な成長パターンに対するモデルの確信度が高いことを意味します。
総合的な解釈
全体トレンドの的確な把握:
青色の平均成長曲線は、広く散らばった黒いデータ点のちょうど中心を滑らかに貫いています。これは、個体差という大きな変動の中から、モデルが集団に共通する基本的な成長パターン(S字カーブ)を抽出できていることを示しています。
モデルの適合度の高さ:
観測されたデータ点は、予測された平均曲線の上下に偏りなく分布しています。もしモデルの仮定(例えばロジスティック曲線という形状)がデータと合っていなければ、ある区間ではデータが系統的に予測線の上側に、別の区間では下側に外れる、といったパターンが見られますが、そのような兆候はありません。
# --- 個体ごとの成長曲線を描画します ---
# 1. conditions引数に渡すためのデータフレームを作成します。
# プロットを描画したいグループの一覧を`group`列に含めます。
make_conditions <- data.frame(group = unique(sim_data$group))
# 2. conditional_effectsを呼び出します。
# effectsに主効果 "day" を、conditionsにグループ一覧を指定します。
ce_plot <- conditional_effects(
fit_brm_nl,
effects = "day",
conditions = make_conditions,
re_formula = NULL # 全てのランダム効果を考慮に含める(デフォルト)
)
# 3. プロットを描画します。
plot_group_fit <- plot(ce_plot, plot = FALSE, ask = FALSE)[[1]] +
geom_point(
data = sim_data,
aes(x = day, y = height),
alpha = 0.3,
inherit.aes = FALSE
) +
labs(
title = "個体ごとの成長曲線と95%信用区間",
x = "日数",
y = "身長"
) +
# 描画領域を個体ごとに分割します
facet_wrap(~group) + theme_bw()
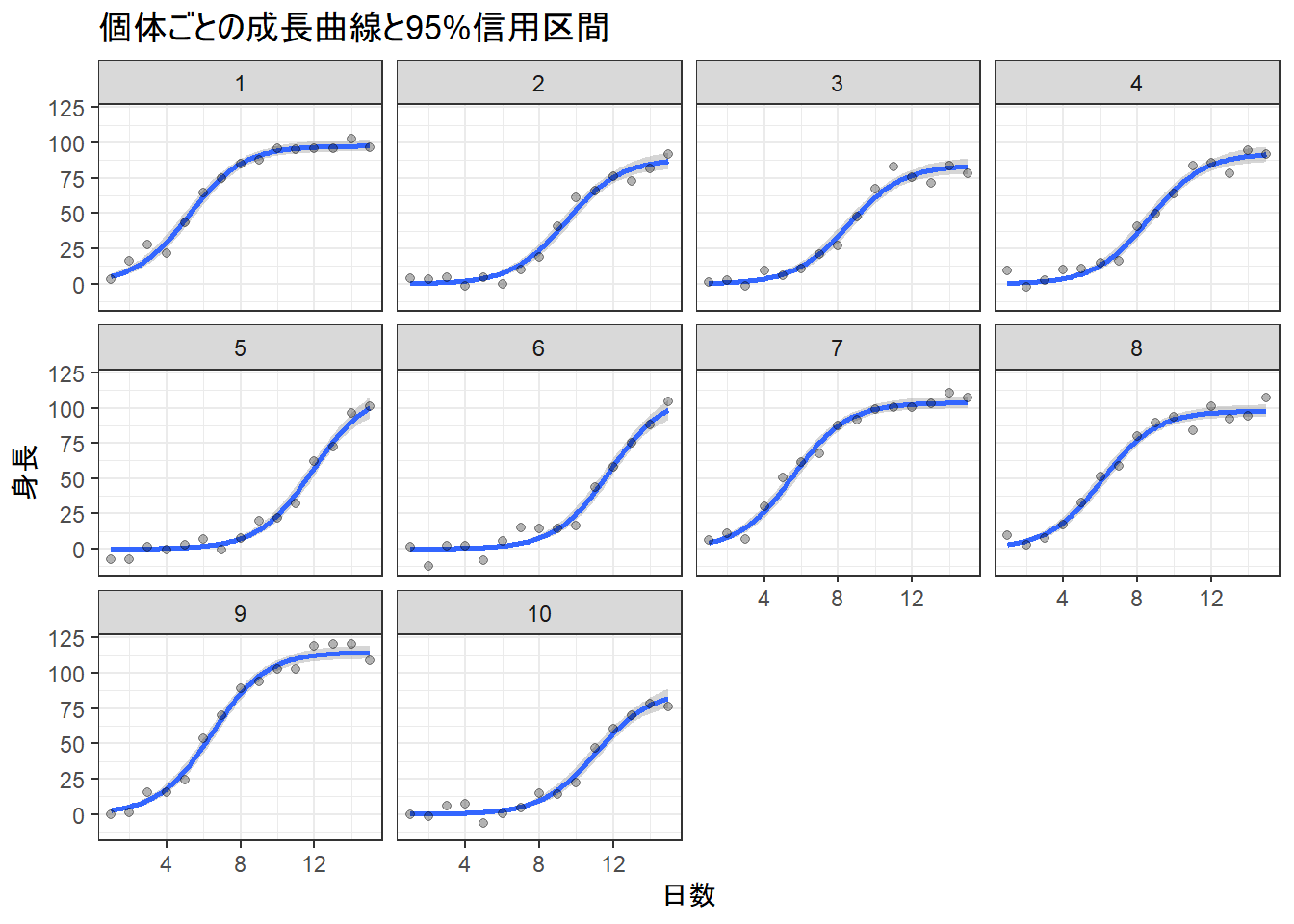
print(plot_group_fit)Figure 2 は、Figure 1 が集団全体の「平均像」を描いたのに対し、Figure 2 は個々の植物が持つユニークな成長パターンを、モデルがどのように捉えたかを明らかにします。
「集団として共通の成長パターン(S字カーブ)を持ちながらも、個体ごとに最終的な身長や成長のタイミングは異なる」という、データの階層構造そのものを視覚化することを目的とします。
1. 10個のパネル
- グラフ全体が、番号1から10が振られた10個の小さなパネルに分割されています。
- 各パネルが、シミュレーションデータにおける一個体 (
group) に対応しています。これにより、個体ごとの振る舞いを直接比較・検討することが可能になります。
2. 各パネル内の要素
各パネルの中には、集団レベルのプロットと同様の要素が描かれていますが、その意味合いは個体に特化しています。
- 黒色の点: そのパネルに対応する特定の一個体から観測された、実際の身長データです。
- 青色の実線: モデルが予測した、その個体のためだけの成長曲線です。この線は、集団全体の平均パラメータと、その個体特有のランダム効果(
Asymとxmidの平均からのズレ)の両方を考慮して描画されています。 - 灰色の帯: その個体の平均予測に対する95%信用区間です。モデルが、その個体の成長曲線をどの程度の不確実性を持って予測しているかを示します。
総合的な解釈
- 個体差の表現
最終身長 (
Asym) の違い:例えば、パネル
1や7の個体は、身長が約100で頭打ち(飽和)になる曲線が描かれています。一方で、パネル5や6の個体は、それよりも高く、100を超えて成長する曲線が予測されています。成長タイミング (
xmid) の違い:パネル
8の個体は、比較的早い段階(日数が8より前)で成長の勢いが最大になっているように見えます。対照的に、パネル10の個体は、成長が本格化するのが少し遅いようです。このように、モデルが
Asym ~ 1 + (1 | group)とxmid ~ 1 + (1 | group)という式で指定された通り、個体ごとに異なるAsymとxmidの値を推定し、それに基づいて個別の予測を行っていることが確認できます。
「情報の共有」による推定
各個体のデータは15点しかありませんが、モデルは滑らかで妥当な成長曲線を推定できています。これは階層モデルの「情報の共有」(Partial Pooling)という特性のおかげです。
各個体の曲線は、その個体自身のデータ点にフィットしようとすると同時に、集団全体の平均的な成長パターンからも情報を「借りて」います。それゆえ、個々のデータのノイズに過剰に適合することなく、全体として、より安定した予測が可能になります。
以上です。